摘要 Abstract
基于最小均方误差重构(LMSER)网络,并利用多尺度表示来预测结合位点。
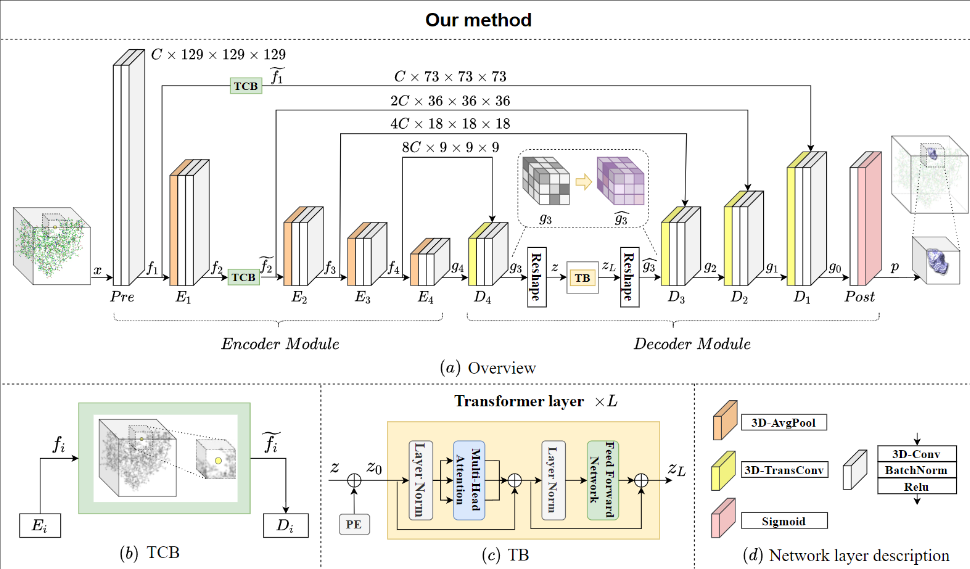
首先,GLPocket使用目标裁剪块(TCB)进行目标预测。TCB从全局表示中选择局部感兴趣的特征进行集中预测。
介绍 Introduction
蛋白质结合位点主要是蛋白质种的深沟或者凹槽的形式(grooves or tunnels)问题研究的意义
problem的困难之处:蛋白质表面凹凸不平,导致出现很多假阳的部位。直径相差比较大,导致预测困难。
介绍相关工作:几何、模板、能量、机器学习、深度学习
根据蛋白质的几何特征预测
从数据库中搜索最相似的蛋白质,分配给查询的蛋白质
最小相互作用能量预测[[13-AutoSite 一个自动化的方法基于配体结合的伪配体预测站点识别是预测的关键配位原子]]
使用物理和化学信息分配和标记口袋点数,用随机森林算法计算绑定分数。
DeepSite使用CNN进行处理预测,但都是局部,没到达体素级别,从蛋白质的三维坐标系出发?[[14-DeepSite 使用 3D 卷积神经网络的蛋白质结合位点预测器]]
论文主要的改进就是在per-voxel的分类上,使用3D U-Net
存在使用整个蛋白质作为输入,另一个是以局部小范围残基作为输入,使用FPocket对多个进行预测,的到每个小范围的候选中心,使用这些候选中心作为输入。建立了解码器到编码器的反馈链路。
肯定了局部区域方法的合理性,但又没有考虑周围环境和全局信息。
是对全局和局部的权衡。
最小均方误差重构(LMSER)比U-Net更好。
论文主要的创新:
使用LMSER来捕捉预测位点
使用蛋白质作为编码阶段的输入,获得全局表示
使用Transformer获取局部肽间的依赖
使用一种目标裁剪块来提取局部
材料和方法 Materials and Methods
四种Datasets scPDB COACH420 HOLOk PDBbind
Architecture of GLPocket
编码模块、解码模块、目标裁剪模块和transformer模块。
使用不同的卷积核编码再加上残差。
从全局中获得局部的映射
我觉得最关键的就是Target Cropping Block (TCB)的作用
获取更多的邻域信息来增强提案的表示能力。
TCB从候选中心的周围作为局部提议,这就是候选中心之间会存在重复的区域。
是对像素属于binding pocket的预测
三个Evaluation Metric
DCA 是预测的口袋中心与最近的配体原子之间的距离。
DCC 是预测口袋中心与标签中心之间的距离。
DVO 是预测的pocket和对应的标签的重叠与它们的体积并集的比率。
结果 Result
通过消融实验验证了TCB和Tranformer的作用。
总结 Conclusion
在本文中,我们提出了一种基于Lmser的网络结构GLPocket,用于捕获对象的多尺度表示以预测结合位点。我们设计了目标裁剪块(TCB),从全局表示中选择感兴趣的局部特征进行目标预测。TCB融合了全局分布信息,使得局部区域表示具有更强的区分性,使预测更加集中和准确。TCB还在不引入额外参数的情况下,将要计算的特征映射的体积大大减少了82%。
问题 Question
多尺度表示学习方法是什么?[[00-PaperQuestion#多尺度表示学习]]
3D UNet是什么? [[00-PaperQuestion#3D UNet]]
最小均方误差重构(LMSER)是什么?[[00-PaperQuestion#LMSER]]
After
Results Understanding
Transformer是有较大的作用的
TCB是一种获得邻域的方式
Methods Cracking
从像素级别进行判断是否合理?和生化性质有一定的区别。
Enhancement or Improvement Ideas
全局和局部结合是提高的一种方式
将模型结合生化性质可能也是一种改进方向
Source Code
def clip(self, data_x, f, condidate_center, a1a2):
center = condidate_center / f
# block_list = []
a1, a2 = a1a2
min_center = (center - a1).int()
max_center = (center + a2).int()
x1, y1, z1 = min_center[0]
x2, y2, z2 = max_center[0]
# print(center)
# print('a1={} a2={}'.format(a1, a2))
# print('x1,y1,z1=', x1, y1, z1)
# print('x2,y2,z2=', x2, y2, z2)
batch_block = data_x[:, :, x1: x2 + 1, y1: y2 + 1, z1:z2 + 1]
return batch_block
其实就是根据中心划分周围区域的一种方式



